



Mot du responsable

L’idée de mettre en commun certains équipements lourds dans le cadre d’une plateforme expérimentale localisée sur le site de la Faculté des Sciences et Technologies à Nancy a émergé peu de temps après la création de l’Université de Lorraine (UL) en 2012 dans le cadre du contrat de plan Etat/Région (CPER).
A partir de 2017, la construction de la plateforme ASIA « Approches fonctionnelles et Structurales des InterActions cellulaires » a connu un nouvel essor. Ainsi quatre unités de recherche (UMR 1136 IAM, UMR 1128 DynAMic, UMR 1434 SILVA et USC INRAE 340 L2A) ont mis en commun des moyens humains et financiers pour la structurer. ASIA a pour objectif de mettre à disposition des technologies de pointes et d’apporter aux chercheurs l’expertise et le savoir-faire associés.
Jean-Michel GIRARDET
Responsable opérationnel

Adresse
Plateforme ASIA
Faculté des Sciences et Technologies
Campus Aiguillettes
BP 70239
54506 Vandœuvre-lès-Nancy Cedex
ASIA est située à l’entrée 1B de la FST, au niveau 6
Responsables
Jean-Michel GIRARDET
Responsable opérationnel
Emilie CLÉMENT
Responsable fonctionnelle
Téléphone
03.72.74.51.66
Actualités :
Publication
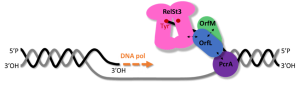
ITC, SEC-MALS et switchSENSE : trois technologies ASIA mises à contribution dans une étude collaborative
Publié le 2025-12-08 09:45Présentation
ASIA regroupe une vingtaine d’équipements pour l’étude de la régulation des activités biologiques et physiologiques des protéines ainsi que l’étude des interactions cellulaires et moléculaires. Les outils disponibles sur la plateforme permettent d’aborder ces questions à différents niveaux.
La plateforme est ouverte aux demandes émanant de laboratoires de recherche académiques ou d’entreprises privées sous la forme de collaborations scientifiques, de mises à disposition d’équipements ou de prestations de service. ASIA accueille notamment des étudiants de différentes formations de l’Université de Lorraine.
En 2018, la plateforme ASIA a mis en place une démarche qualité dans un souci d’amélioration continue. Le 05 mars 2021, ASIA a obtenu le label INFRA+ (3 étoiles) délivré par l’I-site Initiative d’excellence lorraine, gage de qualité pour les utilisateurs.
Domaine d’application
La plateforme ASIA permet de réaliser des analyses du fonctionnement des organismes (arbres, champignons, bactéries). Elle est notamment un support essentiel pour tout ce qui concerne l’étude des technologies blanches de recyclage/déconstruction des bois ou de l’adaptation des écosystèmes forestiers aux climats du futur grâce à leurs microorganismes. ASIA est un appui technologique pour d’autres projets dans des secteurs de recherche visant à identifier et à produire de nouvelles biomolécules fonctionnalisées et vectorisées : Agro-chimie, biocontrôle, agro-alimentaire, cosmétique, pharmaceutique et médical.
La plateforme ASIA a construit sa notoriété autour de sa capacité à pouvoir caractériser les interactions moléculaires régissant les systèmes biologiques.
Publications impliquant la plateforme
Lors de la valorisation de vos projets scientifiques (articles, posters, présentations), pensez à remercier la plateforme ASIA, que le personnel soit co-auteur ou non. Merci d’utiliser la formulation suivante : « This work benefited from the ASIA platform (Université de Lorraine-INRAE; https://a2f.univ-lorraine.fr/asia/). ».
Équipements

switchSENSE heliX+
Le biocapteur heliX+ (et le DRX de première génération) utilise la technologie switchSENSE pour l’étude d’interactions moléculaires en temps réel entre une protéine immobilisée sur une puce à ADN et d’autres partenaires.

LC-MS Analytique et Préparative
L’UHPLC analyse l’activité enzymatique en séparant les produits de réaction avec une haute résolution (haute pression, particules sub-2 µm), permettant d’identifier métabolites, protéines et peptides dans des mélanges complexes.

SEC-MALS
Le MALS couplé au FPLC (ÄKTA) est une technique qui mesure la masse moléculaire des protéines/acides nucléiques en solution. Elle évalue l’homogénéité de l’échantillon, détermine la stœchiométrie des complexes, l’état d’oligomérisation et les changements conformationnels.

Microcalorimètre ITC
L’ITC (MicroCal ITC200) mesure directement la chaleur des interactions biomoléculaires, déterminant affinité (KA), stœchiométrie, enthalpie, entropie et énergie libre (ΔG0) pour un profil thermodynamique complet.

Microscope à épifluorescence
Le microscope motorisé Ti2-E offre un champ unique de 25 mm, autofocus PFS, contrôle complet NIS, fluorescence multi-canal (DAPI, mTurquoise, mNeonGreen, mVenus-Q69H, mScarlet-I), caméra DS-50 60 MP, et enceinte thermostatée ±0,1 °C.

HPTLC et Chromatographie flash
Trois appareils de chromatographie : auto-échantillonneur ATS 4, dérivatisateur Chromajet DS20 et système PuriFlash XS420, assurant application, révélation et séparation automatisées précises pour analyses HPTLC et purification rapide d’échantillons.

FPLC ÄKTA
La FPLC (ÄKTA-purifier, ÄKTA-pure 25L) sépare protéines et acides nucléiques en conditions natives, via colonnes analytiques/préparatives et techniques IMAC, échange ionique, hydrophobe ou SEC, sous 10 bar.

Station bio-informatique
Station de travail Precision 3660 (réf. Sylvain Darnet) : Intel Core i9-13900K, 32 Go DDR5, double SSD NVMe 1 To + HDD 4 To, GPU Intel intégré, Windows 11 Pro, écran incurvé 49″, logiciel XL Stat.

Néphélomètre
Pour le criblage à haut débit (en microplaque) de la croissance microbienne, la solubilité des molécules et la cinétique de liaison des protéines.

Spectrofluorimètre
Un spectromètre à fluorescence permet de mesurer la fluorescence réémise à une longueur d’onde donnée.

Lecteur de microplaques
Le lecteur de microplaque (6 à 384 puits) permet des mesures d’absorbance entre 230-800 nm et de fluorescence entre 203-800 nm. Il permet de réaliser des spectres d’absorption et des spectres d’excitation et d’émission de fluorescence.

Ultracentrifugeuse
Référent : Badreddine DOUZI
Séparation de macromolécules (acides nucléiques, lipoprotéines, etc.) et des organites cellulaires par accélération centrifuge (5000 g à 100 000 g).

Concentrateur sous vide
La concentration par centrifugation sous vide (appareillage miVAC) permet d’éliminer rapidement le solvant organique des échantillons.

Lyophilisateur
La lyophilisation consiste à enlever l’eau d’un produit liquide, pâteux ou solide par sublimation.

Broyeur cryogénique
Grâce au système de refroidissement intégré (azote liquide à -196°C), le bol de broyage est continuellement refroidi à l’azote liquide avant et pendant le broyage. L’échantillon (aliments, bois, boue, morceaux de plantes, semences oléagineuses, sols, textiles…) est ainsi fragilisé et les composants volatils sont conservés.

Soxtherm
L’extraction par solvant sépare rapidement diverses petites molécules de matrices végétales sans filtration. La plateforme ASIA dispose de trois Soxhlets pour des extractions solides-liquides simples et efficaces.

Fibrebag
Détermination des fractions de fibres végétales et alimentaires.

Digitale PCR et qPCR
Réf. Johan Staub : développement d’amorces, détection de mutations ponctuelles, identification d’OGM, agents pathogènes et quantification précise de charges bactériennes, virales ou parasitaires.
Savoir-faire

Préparation et fractionnement des échantillons
Purification et caractérisation de molécules


Etude des interactions moléculaires et microscopie à épifluorescence
Dosages spectroscopiques

Analyse de l’expression génique
et traitement bio-informatique
Personnels

Eric Gelhaye
Directeur scientifique
eric-gelhaye@univ-lorraine.fr

Audrey Nuyttens
Référente LC-MS
audrey.nuyttens@univ-lorraine.fr

Johan Staub
Référent PCR et microscopie
johan.staub@inrae.fr

Badreddine Douzi
Référent ultracentrifugation
badreddine.douzi@inrae.fr
Publications
+60
Enseignants-Chercheurs
+79
Chercheurs
+152
BIATSS et ITA
+300
Publications







